Hallo,
mir wurde aufgetragen, meine Versuchsergebnisse statistisch abzusichern. Leider kenne ich mich bisher relativ wenig mit statistischen Tests aus und hoffe ihr könnt mir weiterhelfen.
Ich denke, früher oder später werde ich wohl noch einen Statistikkurs besuchen müssen =)
Ich habe untersucht, ob sich ein bestimmter Typ von Retroposons (DNA-Elemente, die Kopien von sich selbst 'zufällig' an einer anderen Stelle wieder in die DNA einbauen) in der DNA GC- oder AT-reiche Regionen bevorzugen.
(GCAT = 4 DNA Basen).
Dazu habe ich jeweils einen Bereich von etwa 2000 Basenpaaren um die Einbaustelle des Retroposons untersucht und den GC-Gehalt errechnet. Ich habe also für jedes Retroposon einen Prozentwert, wieviele G und C Basen sich in der Umgebung befinden. Insgesamt habe ich um die 10.000 Retroposons untersucht.
Zusätzlich habe ich 10.000 zufällige Positionen im Genom ausgewählt und von diesen Positionen ebenfalls 2000 Basenpaar lange DNA-Sequenzen extrahiert. Hier habe ich ebenfalls den GC-Gehalt errechnet.
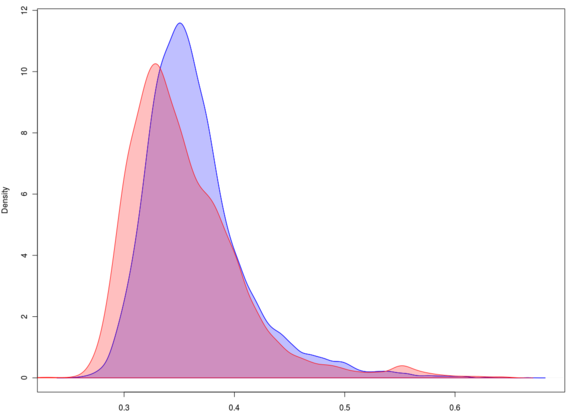
Die Ergebnisse habe ich in R geplottet (densityplot s.Screenshot). Die blaue Kurve zeigt den GC-Gehalt der Retroposon-Umgebung, die rote Kurve den GC-Gehalt der Zufallsregionen.
In Retroposon-Umgebung ist der GC-Gehalt also leicht höher als der durchschnittliche GC-Gehalt im Genom.
Wie kann ich dieses Ergebnis nun statistisch untermauern / widerlegen? Welche Tests sind hier sinnvoll?
Bin um jede Hilfe dankbar =/
LG
Philipp
Welcher statistische Test?
Thema bewerten: 




 • 2 Beiträge
• Seite 1 von 1
• 2 Beiträge
• Seite 1 von 1
Welcher statistische Test?
- Dateianhänge
-
- screenshot3 .resized.png (23.16 KiB) 814-mal betrachtet
- knabberknut
- Einmal-Poster

- Beiträge: 1
- Registriert: Mi 2. Nov 2011, 15:45
- Danke gegeben: 0
- Danke bekommen: 0 mal in 0 Post
Re: Welcher statistische Test?
Bei der großen Zahl an Fällen ist es fast egal - der Test wird positiv ausfallen. In dem Fall also 'keep it simple' und mach einfach nur einen Rangsummen-Test der den GC-Gehalt der einen 10.000 Genomorte mit dem GC-Gehalt der anderen 10.000 Genomorte vergleicht. Wenn der Dir aber aus irgendwelchen Gründen nicht passt käme auch ein Permutationstest in Frage. Da es ohnehin signifikant wird, würde ich mir die Diskussion um t-Test und Normalverteilung und geringe Bedeutung der Nicht-Normalverteilung bei großen Zahlen sparen.
JMTC,
Bernhard
PS: Rangsummentest mit wilcox.test(), Permutationstest mit perm.test aus dem package exactRankTests.
JMTC,
Bernhard
PS: Rangsummentest mit wilcox.test(), Permutationstest mit perm.test aus dem package exactRankTests.
----
`Oh, you can't help that,' said the Cat: `we're all mad here. I'm mad. You're mad.'
`How do you know I'm mad?' said Alice.
`You must be,' said the Cat, `or you wouldn't have come here.'
(Lewis Carol, Alice in Wonderland)
`Oh, you can't help that,' said the Cat: `we're all mad here. I'm mad. You're mad.'
`How do you know I'm mad?' said Alice.
`You must be,' said the Cat, `or you wouldn't have come here.'
(Lewis Carol, Alice in Wonderland)
- bele
- Schlaflos in Seattle

- Beiträge: 5944
- Registriert: Do 2. Jun 2011, 23:16
- Danke gegeben: 16
- Danke bekommen: 1405 mal in 1391 Posts
Thema bewerten: 




 • 2 Beiträge
• Seite 1 von 1
• 2 Beiträge
• Seite 1 von 1
Wer ist online?
Mitglieder in diesem Forum: 0 Mitglieder und 5 Gäste